Monocle3單細(xì)胞擬時(shí)序軌跡方法新升級(jí),還不快來(lái)試試?
Monocle3背景簡(jiǎn)述
在外界刺激或者發(fā)育過(guò)程中,不同細(xì)胞的功能會(huì)發(fā)生不同的變化,從而導(dǎo)致細(xì)胞的表達(dá)量呈現(xiàn)出差異性。根據(jù)每個(gè)細(xì)胞的基因表達(dá)情況對(duì)這些細(xì)胞進(jìn)行排序,能夠進(jìn)一步推斷出細(xì)胞發(fā)育的軌跡。
Junyue Cao(曹俊越)等[1]在進(jìn)行小鼠的發(fā)育生物學(xué)研究時(shí),由于數(shù)據(jù)缺少一些祖先的狀態(tài),且已知的一些細(xì)胞類型來(lái)自多個(gè)轉(zhuǎn)錄不同的譜系,因此為了克服這些限制,同時(shí)將擬時(shí)序方法擴(kuò)展到數(shù)百萬(wàn)個(gè)細(xì)胞,于是開(kāi)發(fā)了以PAGA算法[2]為藍(lán)本的新方法,即Monocle3。該方法因其高效的運(yùn)算速度和美觀的可視化,迅速登上了單細(xì)胞擬時(shí)序分析的舞臺(tái),并已在《Nature》雜志上發(fā)表。
Monocle3軟件介紹
相比以往熟知的Monocle2,Monocle 3經(jīng)過(guò)了重新設(shè)計(jì),使之可以用于分析大型、復(fù)雜的單細(xì)胞數(shù)據(jù)集。Monocle 3的核心算法具有高度可擴(kuò)展性,可以處理數(shù)百萬(wàn)個(gè)細(xì)胞。軟件主要可以執(zhí)行三類分析:1)對(duì)細(xì)胞聚類、分群和計(jì)數(shù);2)構(gòu)建單細(xì)胞軌跡;3)差異表達(dá)分析。Monocle 3在R語(yǔ)言(v4.1.0或更高版本)環(huán)境中運(yùn)行,同時(shí)需要R包Bioconductor(v3.14或更高版本)和monocle3(v1.2.7或更高版本)來(lái)訪問(wèn)最新功能。
Monocle3原理介紹
1. 使用UMAP降維
統(tǒng)一流形逼近和投影 (UMAP) 是一種降維技術(shù),Monocle3首先將數(shù)據(jù)投影到一個(gè)低維空間,基于黎曼幾何和代數(shù)拓?fù)涞乃惴▉?lái)執(zhí)行降維和數(shù)據(jù)可視化。與t-SNE相比,UMAP的可視化更具有競(jìng)爭(zhēng)力,最顯著的是提高了運(yùn)行速度并更好地保存了數(shù)據(jù)的全局結(jié)構(gòu)。結(jié)果發(fā)現(xiàn),UMAP的計(jì)算效率顯著地加快了對(duì)小鼠胚胎數(shù)據(jù)的分析。UMAP在大約3個(gè)CPU小時(shí)內(nèi)完成了處理200萬(wàn)個(gè)單元格的數(shù)據(jù)集,而t-SNE花費(fèi)了超過(guò)64個(gè)CPU小時(shí)。當(dāng)然,Monocle3也可以直接導(dǎo)入Seurat的降維結(jié)果進(jìn)行分析。
2. 將細(xì)胞劃分為不連續(xù)的軌跡
細(xì)胞的分化總是具有連續(xù)性和離散性的。當(dāng)數(shù)據(jù)量很大時(shí),Monocle2可能會(huì)錯(cuò)誤地把本來(lái)不在同一個(gè)軌跡上的細(xì)胞認(rèn)為是在同一個(gè)軌跡上,而PAGA(基于分區(qū)的圖抽象方法,Partition-based graph abstraction)方法則解決了這個(gè)困難。
3. 軌跡圖學(xué)習(xí)
Monocle3在相同的低維空間進(jìn)行軌跡圖學(xué)習(xí),來(lái)表示細(xì)胞在發(fā)育過(guò)程中可能采取的路徑。對(duì)此,Monocle3使用了一個(gè)基于SimplePPT算法的圖嵌入過(guò)程。另外,Monocle3還有幾點(diǎn)升級(jí):1)直接在UMAP空間(默認(rèn)為三維的)中繪制軌跡主圖,避免直接處理成千上萬(wàn)個(gè)單細(xì)胞數(shù)據(jù);2)能夠平滑和細(xì)化主圖以排除小分支,從而消除噪聲分支;3)適用于環(huán)路軌跡,而不要求分化軌跡一定是樹(shù)形結(jié)構(gòu)。
4. 計(jì)算偽時(shí)間
為了計(jì)算細(xì)胞級(jí)別的偽時(shí)間,Monocle3開(kāi)發(fā)了一種適用于具有數(shù)百萬(wàn)細(xì)胞數(shù)據(jù)集的投影策略。該策略的原理是,以主圖為指導(dǎo),在所有單元上構(gòu)造一個(gè)圖ψ,然后通過(guò)計(jì)算每個(gè)細(xì)胞與選擇的一個(gè)根或多個(gè)根的距離作為偽時(shí)間。Monocle3是一個(gè)半監(jiān)督的擬時(shí)序分析,因此需要通過(guò)生物學(xué)背景來(lái)選擇合適的起始點(diǎn)。
5. 識(shí)別表達(dá)基因
為了識(shí)別在發(fā)育軌跡中表達(dá)變化不同的基因,Monocle3運(yùn)用了一種常用于分析空間數(shù)據(jù)的統(tǒng)計(jì)檢驗(yàn)。Moran’s I統(tǒng)計(jì)量(莫蘭指數(shù))是一個(gè)多向和多維空間自相關(guān)的度量。該統(tǒng)計(jì)量通過(guò)最近鄰圖編碼數(shù)據(jù)點(diǎn)之間的空間關(guān)系,使其特別適合于分析大型單細(xì)胞轉(zhuǎn)錄組數(shù)據(jù)集。Moran指數(shù)的范圍在-1~1之間,0代表此基因沒(méi)有空間共表達(dá)效應(yīng),1代表此基因在空間距離相近的細(xì)胞中表達(dá)值高度相似,小于0的Moran指數(shù)一般都沒(méi)有統(tǒng)計(jì)學(xué)意義。
Monocle3分析步驟
1. 數(shù)據(jù)導(dǎo)入
首先將Seurat對(duì)象轉(zhuǎn)變成軟件可以識(shí)別的cds對(duì)象。將Seurat對(duì)象拆分成三個(gè)模塊:基因的表達(dá)矩陣(稀疏矩陣)、metadata細(xì)胞屬性信息、基因?qū)傩孕畔ⅰ?/p>
生成對(duì)應(yīng)的三個(gè)模塊數(shù)據(jù):
data <- GetAssayData(M_3h, assay = ''RNA'', slot = ''counts'') ?#細(xì)胞的表達(dá)矩陣new_pd = M_3h@meta.data #meta信息fData <- data.frame(gene_short_name = row.names(data), row.names = row.names(data)) #基因?qū)傩孕畔?/p>
使用三個(gè)模塊數(shù)據(jù)創(chuàng)建cds對(duì)象:
cds <- new_cell_data_set(expression_data=data,cell_metadata=new_pd,gene_metadata= fData)
2. 數(shù)據(jù)預(yù)處理
數(shù)據(jù)預(yù)處理包括對(duì)數(shù)據(jù)進(jìn)行歸一化、標(biāo)準(zhǔn)化、去除批次、PCA降維等,并設(shè)置一個(gè)PCA維度(這里設(shè)置為10),可以用plot_pc_variance_explained()函數(shù)來(lái)展示維度。
cds <- preprocess_cds(cds, num_dim = 10)
3. 非線性降維和可視化
一般使用UMAP或t-SNE進(jìn)行非線性降維和可視化。Moncle3更推薦使用UMAP方法,處理單細(xì)胞數(shù)據(jù)更快。
cds <- reduce_dimension(cds,preprocess_method = ''PCA'',reduction_method = ''UMAP'')
4. 聚類分析
聚類分析可以判別哪些細(xì)胞是來(lái)自一個(gè)祖先(一個(gè)發(fā)育軌跡上)。
cds <- cluster_cells(cds)
5. 擬時(shí)序分析
擬時(shí)序分析用來(lái)找出每個(gè)群內(nèi)的細(xì)胞發(fā)育軌跡。利用擬時(shí)序分析的結(jié)果可以繪制擬時(shí)序圖。
cds <- learn_graph(cds) #軌跡推斷plot_cells(cds, color_cells_by = "partition", cell_size = 0.5)
6. 選擇合適的起點(diǎn)
可以通過(guò)算法計(jì)算起始點(diǎn),也可以通過(guò)手動(dòng)選擇起始點(diǎn),考慮到生物學(xué)意義,推薦手動(dòng)設(shè)置起點(diǎn)。
cds <- order_cells(cds, root_cells = root.cell) #選擇對(duì)應(yīng)的細(xì)胞群作為起點(diǎn)
7. 下游分析
下游分析包括差異基因分析、單個(gè)基因分析(判斷基因的激活順序)等。
Monocle3結(jié)果展示
圖1 | 擬時(shí)序UMAP圖
圖片說(shuō)明:圖中每個(gè)點(diǎn)代表一個(gè)細(xì)胞,具有相似狀態(tài)的細(xì)胞被聚到一起,顏色標(biāo)注不同的細(xì)胞群。黑色線條表示分化軌跡。黑色圓圈表示樹(shù)干節(jié)點(diǎn)(branch nodes),表示從這個(gè)點(diǎn)開(kāi)始,細(xì)胞可以有多個(gè)結(jié)局;灰色圓圈表示分化樹(shù)葉節(jié)點(diǎn)(leaf nodes),即細(xì)胞的不同結(jié)局。
圖2 | 擬時(shí)序軌跡圖
圖片說(shuō)明:顏色由紫色到黃色,表示分化的時(shí)間由早及晚。數(shù)字表示分化的過(guò)程,數(shù)字越小,表示分化的時(shí)間越早。另外,如果自己指定的起始點(diǎn),無(wú)法直接訪問(wèn)到某一部分細(xì)胞,這部分細(xì)胞就有無(wú)窮偽時(shí)間,用灰色表示。
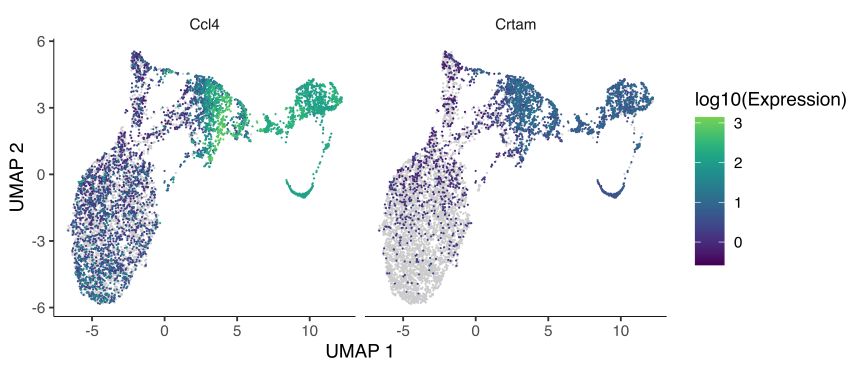
圖3 | 擬時(shí)序基因表達(dá)featureplot圖
圖片說(shuō)明:顏色的深淺表示基因表達(dá)量的高低,灰色部分表示該基因沒(méi)有在這些細(xì)胞內(nèi)進(jìn)行表達(dá)。
圖4 | 擬時(shí)序基因表達(dá)曲線圖
圖片說(shuō)明:每個(gè)點(diǎn)的顏色表示不同的細(xì)胞群,橫軸從左到右表示時(shí)間的早晚。從圖中可以看出基因隨時(shí)間發(fā)展的表達(dá)量變化。
今天我們對(duì)Monocle3的簡(jiǎn)介就到此,不知道您有沒(méi)有g(shù)et到這款好用便捷又快速的軟件呢?有需要的老師趕快試試吧!
參考文獻(xiàn):
[1] Cao, J., Spielmann, M., Qiu, X. et al. The single-cell transcriptional landscape of mammalian organogenesis. Nature 566, 496-502 (2019).?
https://doi.org/10.1038/s41586-019-0969-x
[2] Wolf, F.A., Hamey, F.K., Plass, M. et al. PAGA: graph abstraction reconciles clustering with trajectory inference through a topology preserving map of single cells. Genome Biol 20, 59(2019).?
https://doi.org/10.1186/s13059-019-1663-x
猜你想看
1、Plant Physiol l 西北農(nóng)林科技大學(xué)李強(qiáng)王保通研究團(tuán)隊(duì)發(fā)現(xiàn)谷胱甘肽S-轉(zhuǎn)移酶增強(qiáng)小麥抗白粉病的分子機(jī)制
2、Cell Discov | 單細(xì)胞轉(zhuǎn)錄組測(cè)序助力解析圓錐角膜的發(fā)病機(jī)制
3、Cell Discov | 單細(xì)胞多組學(xué)測(cè)序助力解析腎透明細(xì)胞癌的發(fā)病機(jī)制
4、Stem Cell Res Ther | 單細(xì)胞轉(zhuǎn)錄組測(cè)序助力解析hUCB-MSCs治療宮腔粘連疾病機(jī)制研究