NGS體細胞突變檢測兩三事
二代測序(Next Generation Sequencing, NGS)是一個強大的功能平臺,它可以同時給數以萬計的DNA分子進行測序。由于這種可以多個樣本同時測序的能力,在個性化醫療、遺傳疾病和臨床診斷等方面,二代測序也就是高通量測序開創了革命性的領域。但是,對于癌癥分子診斷、治療和監測等需要檢測低頻基因變異的領域,二代測序錯誤是一種可能影響變異檢測結果的關鍵混雜因素。
我們常用的體細胞突變(somatic mutation)檢測軟件包括:mutect2、strelka、varscan等。那么對于突變檢測,我們還有哪些要注意呢?
一個典型的NGS工作流程包含多個步驟,包括樣品處理、DNA提取、PCR擴增、上機測序。這些步驟中的每一步都可能引入錯誤。例如,樣品處理過程中的DNA損傷可引起C>A/G>T錯誤,DNA提取或片段化過程中甲基化胞嘧啶自發脫氨成尿嘧啶可引起C>T/G>T錯誤,另外,目標富集PCR及測序步驟也會引入一些錯誤。
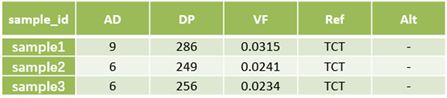
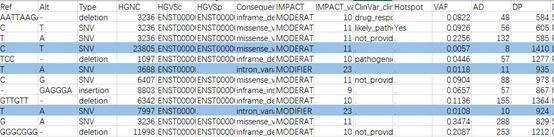
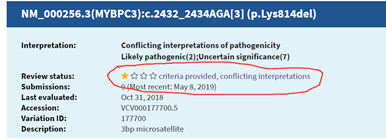
在體細胞突變檢測中,你是否會注意一些異常現象,偶爾會發現一些突變被質控掉了,但這是個例嗎?回顧數據會發現,咦?好像同批次個別樣本也會有這個突變?例如序列CAGCCGCATCCACCGGTAGCTCTTCTTCTTCTTGCGCT,紅色是deletion區域。當然,這個突變被質控掉了,變異檢測軟件標記的str_contraction。但你是否有看數據的習慣呢?仔細觀察突變附近的基因組序列發現該缺失有四個重復單元,是短串聯重復區域(STR區域)的特征,這與注釋也相符。我們知道,STR檢測一般會有滑脫現象,等位基因數值越大(重復的單元數越多)滑脫越多,而等位基因數小的理論上滑脫的可能性很低。
表1 異常體細胞突變示例
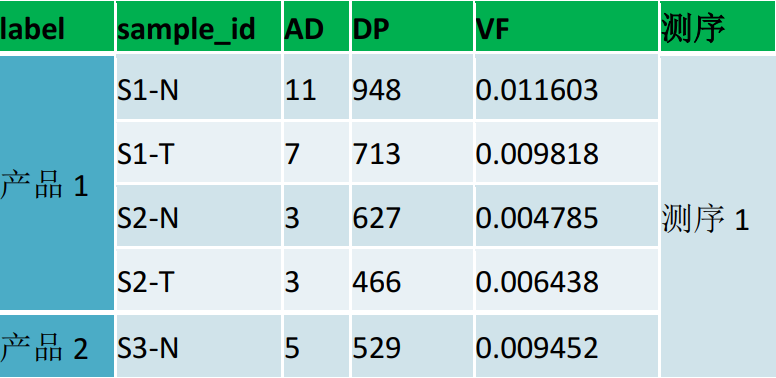
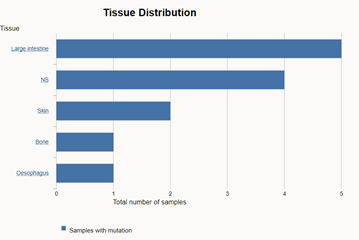
并且對于不同的基因組版本都存在這個現象。另外,由于該突變在同一批數據的多個樣本中發現,突變頻率較低。到底是捕獲探針(原材料)的問題呢?還是STR區域的問題?還是NGS手段的問題?重新設計多重PCR引物,對樣本進行檢測發現與捕獲探針一致,對NGS各個平臺、公司的數據回溯分析,包括腫瘤樣本和正常樣本,發現超過20%的數據(正常對照)包含這個位點,而且頻率都比較低(1%上下浮動)。
表2 不同公司不同產品不同測序平臺分析
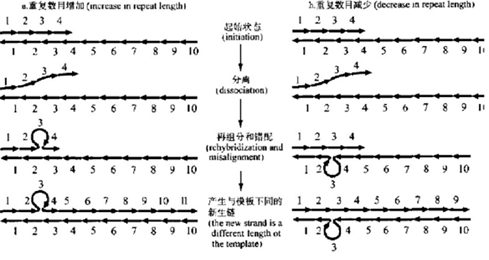
因此判定這個位點是NGS系統性的誤差。那就要考慮基因STR區域本身的特點造成的,STR到底是個什么東東呢,為什么會有這種詭異的影響?讓我們來解開它神秘的面紗。短串聯重復序列(Short tandem repeats, STR),又稱微衛星DNA(Microsatellite DNA),通常是基因組中由1~6個堿基單元組成的一段DNA重復序列,是廣泛存在于真核生物基因組中的核苷酸重復序列。由于重復單位及重復次數不同,構成了STR基因座的遺傳多態性。每個STR由結構包括:具有短重復單元的核心區、核心區兩側保守的側翼區。
圖1 STR結構示意圖
圖2 微衛星的滑鏈錯配突變模型
如果按照默認的軟件指標對變異進行過濾可能會造成假陽或假陰,也就是造成檢測錯誤,而且錯誤還可能來源于基因組組裝錯誤(干試驗+濕實驗)。所以軟件質控并不是萬用的,對于體細胞突變檢測,即使是配對樣本,我們也需要PON,而對于假陰性,我們同樣需要累積(包括cosmic公共數據庫使用和內部積累),平臺的數據累積才是最合適的質控。臨檢的分析雖沒有動植物科研的多樣化手段和各種美圖顯得復雜,但是我們對準確度的要求非常高,開展檢測一定要慎重。
-
科技前沿

-
科技前沿